sobereva 发表于 2023-9-30 05:08
利用AMBER的leap产生拓扑文件,然后再通过acpype等程序转成GROMACS的拓扑文件
huashang 发表于 2023-9-30 12:22
感谢老师的回答,但是我在将pdb文件导入amber的tleap模块中时,又出现新的问题。
Frozen-Penguin 发表于 2023-9-30 13:20
出现 Created a new atom 说明PDB中有力场中没有的原子,出现 Added missing heavy atom 说明PDB中缺少了 ...
huashang 发表于 2023-9-30 21:41
感谢,在按照GLYCAM力场的残基修改完pdb文件后,已经不会出现原子错误了,但是最后生成top文件的时候,又 ...
userrr 发表于 2025-3-13 11:29
你好,博主,我现在也遇到和你一样的问题,我也想用gromacs模拟糖的力场,但是我用ambertools的时候转化我 ...
huashang 发表于 2025-3-15 22:36
我不知道你所说的糖是否是纤维素?如果是纤维素的话,建议使用cellulose-builder来构建纤维素,产生的纤 ...
userrr 发表于 2025-3-18 08:48
你好,博主,我用的就是cellulose构建的纤维素,也产生的pdb文件,但是我用ambertool的时候它的残基好像 ...
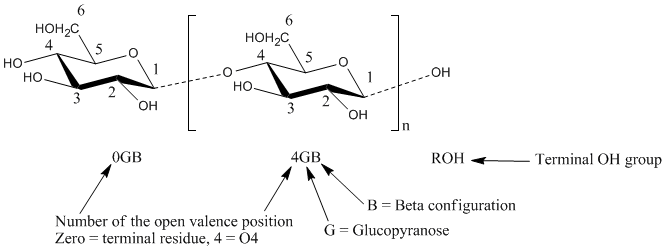
huashang 发表于 2025-3-21 17:12
cellulose-builder生成的纤维素不能直接使用,你需要修改其中残基名,你看看这张图
| 欢迎光临 计算化学公社 (http://bbs.keinsci.com/) | Powered by Discuz! X3.3 |