直接在线访问链接:https://molstar.szbl.ac.cn/viewer
安装安装包:
点击链接进入Qbics-MolStar网站界面:https://molstar.szbl.ac.cn/download/
点击如图所示位置执行下载:
使用Qbics-MolStar有两种方式:
直接在线访问链接:https://molstar.szbl.ac.cn/viewer
安装安装包:
点击链接进入Qbics-MolStar网站界面:https://molstar.szbl.ac.cn/download/
点击如图所示位置执行下载:

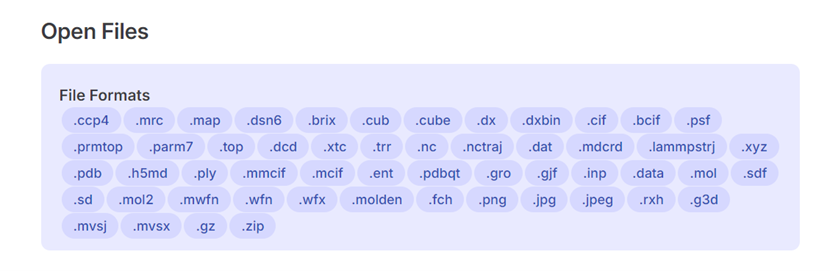
当前支持文件格式包括:.top,.cif,.gjf,.inp,.mol,.xyz,.pdb,.mwfn,.mol2等等,具体介绍详见https://molstar.szbl.ac.cn/docs/use/。
用户可以通过两种方式实现已知分子坐标的可视化(以.pdb为例):
直接拖拽文件至Qbics-MolStar界面,即可实现可视化:
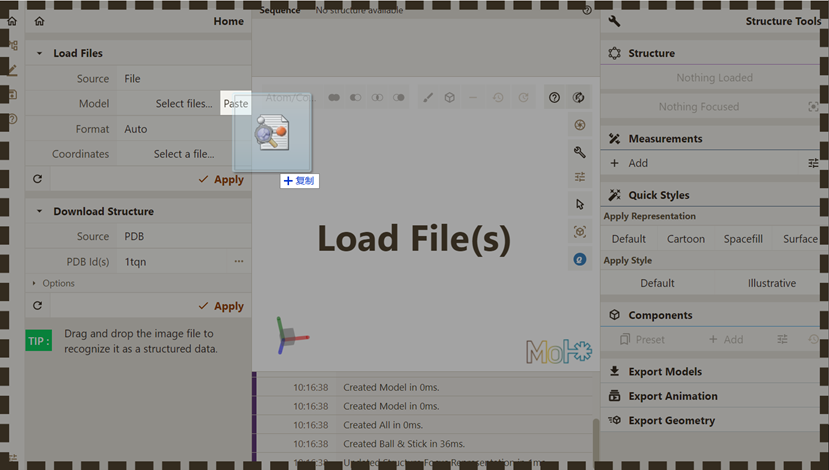
选择保存在本地的文件:
点击下方红框按钮,选择希望可视化的文件。
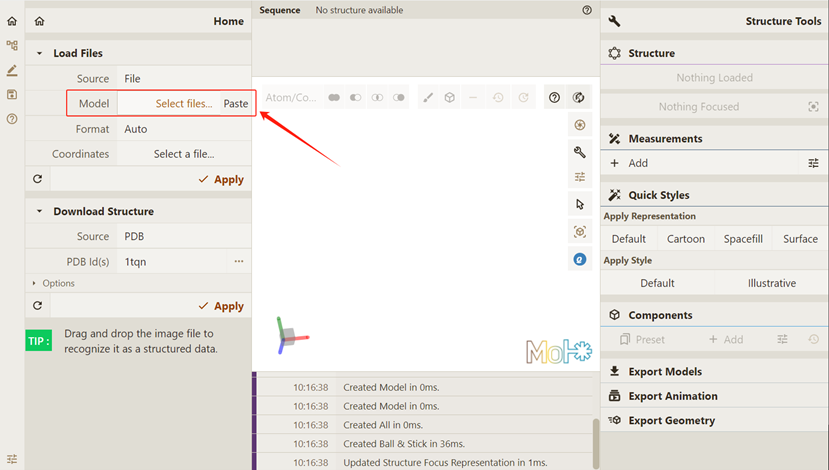
双击选中的文件(此处为c60.pdb)使其加载至Qbics-MolStar:
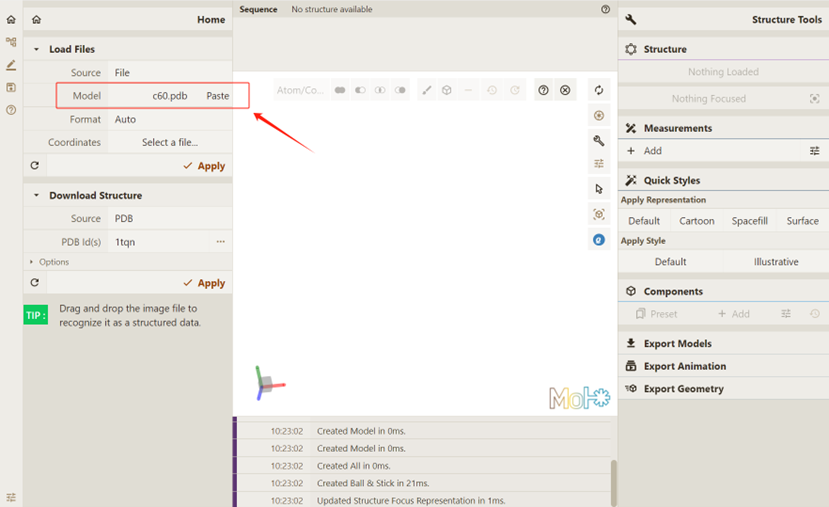
点击 Apply,使Qbics-MolStar开始渲染体系,实现可视化:
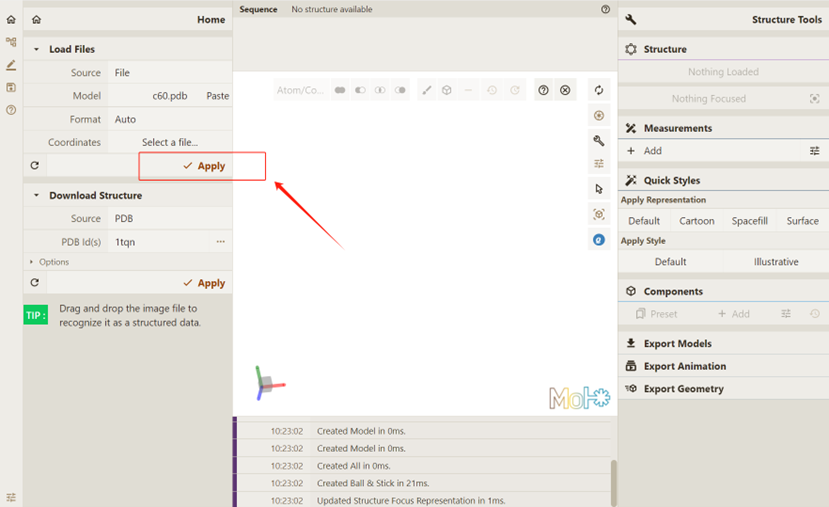
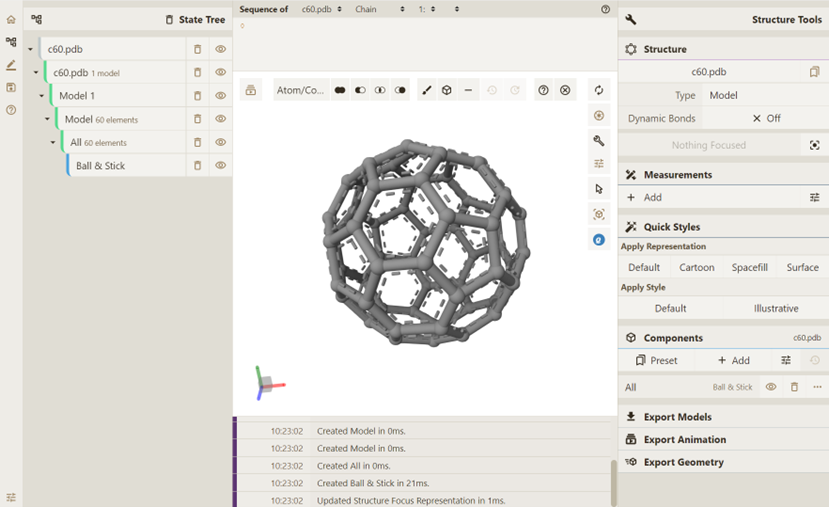
效果如下:
Qbics-MolStar可以从多种途径下载分子坐标并渲染可视化。以下以PDB为例。
在Qbics-MolStar中键入PDB Id,如 6AP4:
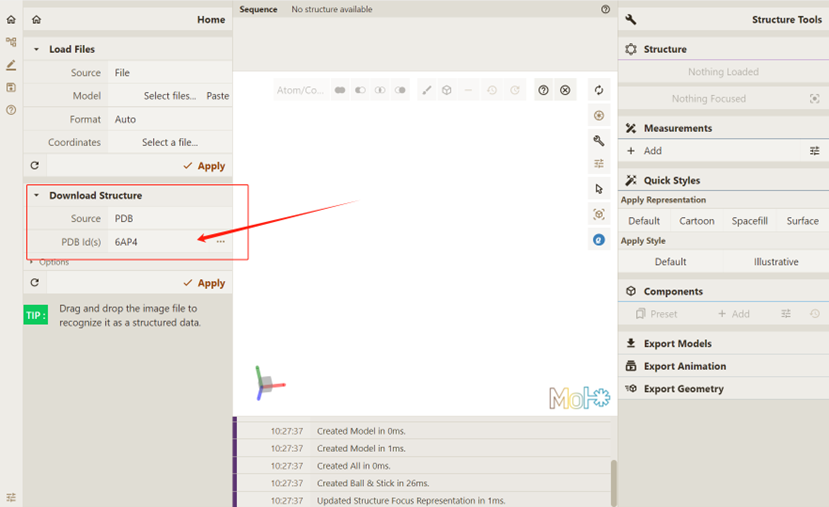
点击 Apply,要求Qbics-MolStar实现可视化:
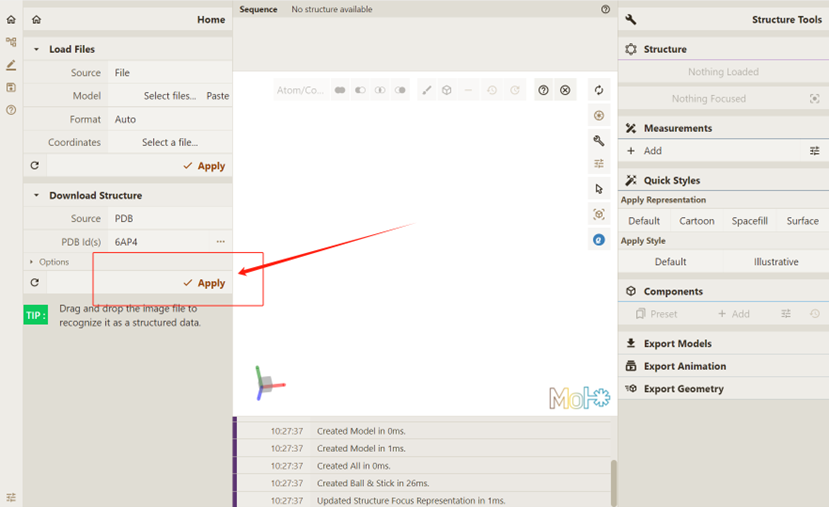
效果如下:
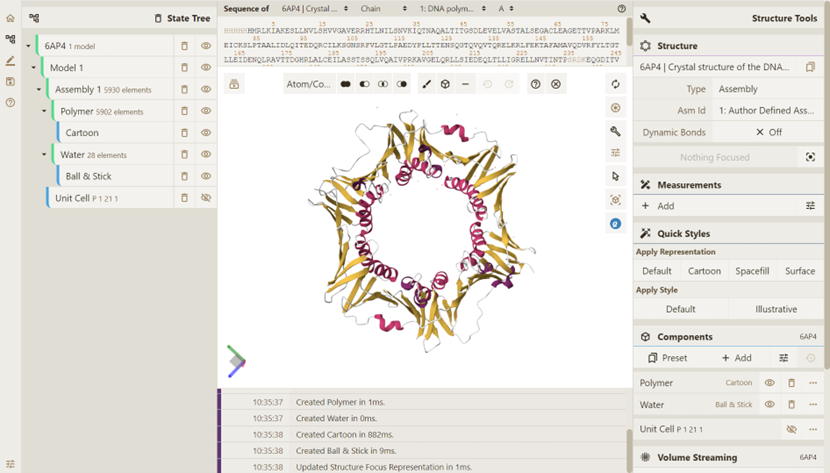 1.4. 分子可视化:SMILES代码
1.4. 分子可视化:SMILES代码Qbics-MolStar还可以根据SMILES, PubChem等导入数据。我们选择SMILES代码作为另一可视化示例。
修改Source为SMILES而非默认PDB:
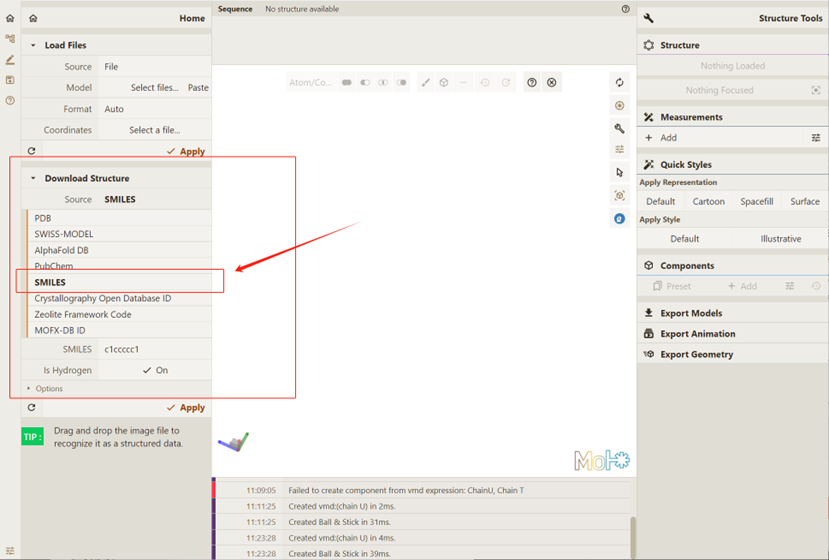
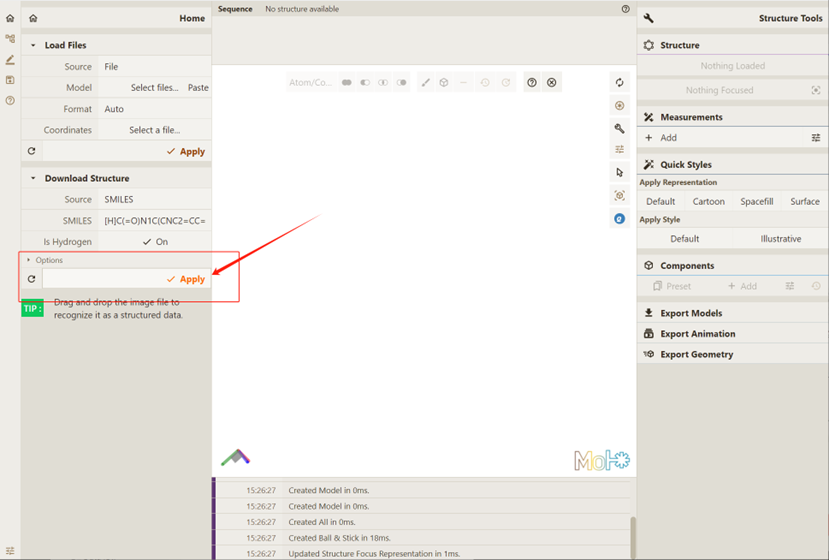
作为本次尝试的SMILES示例,键入要求填写SMILES的位置: [H]C(=O)N1C(CNC2=CC=C(C=C2)C(=O)N[C@@H](CCC(O)=O)C(O)=O)CNC2=C1C(=O)NC(N)=N2:
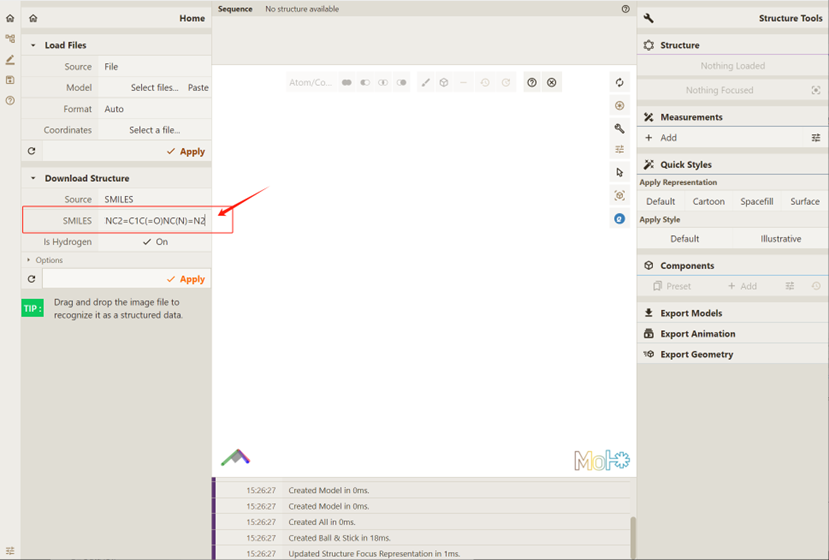
点击 Apply,要求Qbics-MolStar实现可视化:
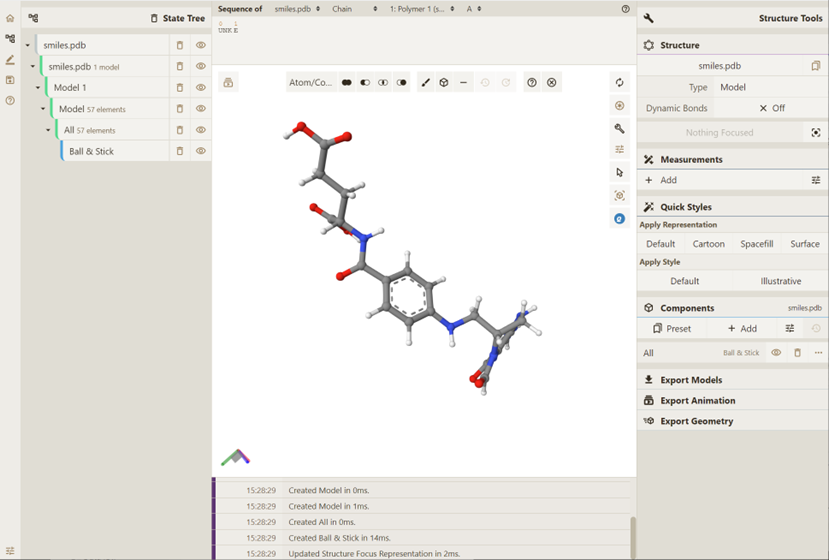
效果如下:
ggdh 发表于 2025-9-16 15:46
快点搞个api!支持用户编写插件!

slxc920113 发表于 2025-9-17 14:09
试用了一下,现在手动建模的时候,片段拼接的二面角还是不太合理,没有像GaussView那样默认是一个位阻较小 ...
 希望开发越来越好!
希望开发越来越好!hdhxx123 发表于 2025-9-18 12:23
这是真的优秀啊!建议吸取VMD的色彩光照材质和脚本自由度的优点,再把内置个xtb的扫把软件希望开发越 ...
greatzdk 发表于 2025-9-19 10:16
问个问题。我是网页点开直接用的。
画好了 用O替代C 就出现如图的问题了。是我哪里用的不对嘛?
coolrainbow 发表于 2025-9-19 14:21
你再实验一下?我这里没有发现问题
mcard 发表于 2025-9-23 20:39
之前研究过mol*,虽然他是模块化的,但感觉文档不是很全,改起来少不了源码来回翻。我记得他源码里面实现了 ...
greatzdk 发表于 2025-9-24 12:55
我试了试 别的画法 好像问题不大。但是 又发现了新的问题,我觉得总归是哪里不太对 你看看

迎迎 发表于 2025-9-24 21:37
今天才下载,发现下载之后第一次点开提示有更新,点了确定,然后有一个进度条,很快就更新完成,点了完成更 ...



Assiduitas 发表于 2025-9-30 11:19
大佬,如果用你们这软件作图。怎么引用你们的成果呢?有已发表的论文吗?.
有个疑惑:参考sob老师的帖子进 ...
Assiduitas 发表于 2025-9-30 11:19
大佬,如果用你们这软件作图。怎么引用你们的成果呢?有已发表的论文吗?.
有个疑惑:参考sob老师的帖子进 ...
Huschein 发表于 2025-10-15 09:38
请问是否有第一作者和通讯作者的联系方式呢?我在想是否可以私下交流一下?
Assiduitas 发表于 2025-9-30 11:19
大佬,如果用你们这软件作图。怎么引用你们的成果呢?有已发表的论文吗?.
有个疑惑:参考sob老师的帖子进 ...
influenza 发表于 2025-9-25 10:45
请问能打开高斯运行任务的输出文件吗? xxx.log 我这里打开就软件卡住只能关掉。
| 欢迎光临 计算化学公社 (http://bbs.keinsci.com/) | Powered by Discuz! X3.3 |